Nature Machine Intelligence|DeepDirect:基于对抗学习指导突变以增强蛋白质亲和力的设计框架
在生物医学和生物技术的发展中,尤其在药物设计、治疗性蛋白质的开发和疫苗构建方面,蛋白质工程扮演了至关重要的角色。蛋白质工程的核心挑战之一是理解和预测蛋白质的结构、功能与序列之间的复杂相互作用。
近年来,随着生物信息学和计算生物学的进步,蛋白质序列到结构以及功能到序列的预测取得了显著进展。这些突破为深入理解蛋白质的生物学功能和进行定向蛋白质设计提供了强大的工具。
然而,在药物设计和蛋白质工程中,预测蛋白质或蛋白质复合体因突变而导致的亲和力变化是一个特别具有挑战性的问题,能够准确预测突变如何影响蛋白质亲和力的技术,将对疾病治疗和生物医学研究产生重要影响。
2月28日,中国科学院深圳先进技术研究院先进计算与数字工程研究所研究员、深圳理工大学(筹)计算机科学与控制工程学院杰出教授李金艳研究团队在Nature Machine Intelligence上发表了题为“Generating mutants of monotone affinity towards stronger protein complexes through adversarial learning”的研究文章。该文章介绍了一种创新的模型DeepDirect,旨在通过对抗学习生成的突变方法,显著改变蛋白质亲和力的方向,为蛋白质生物工程、药物设计等领域提供新的思路。
团队展示了如何利用深度学习生成蛋白质突变,以优化其功能和亲和力,特别是在研究SARS-CoV-2 Omicron变种时的应用。这项工作不仅提供了一种高效的蛋白质改造策略,还为理解蛋白质动态和疾病进化提供了新的视角,对未来的生物医药研究具有重要意义。

论文截图
DeepDirect模型由蛋白质序列突变生成器、两个鉴别器和一个蛋白质复合体亲和力变化预测器三个主要部分组成。突变生成器是DeepDirect架构中最重要的部分(见图1和图2a)。
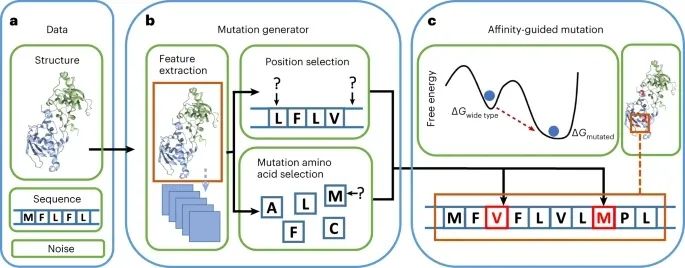
DeepDirect突变生成器的概述(图1)
生成器的输入需要三种类型的数据:蛋白质的氨基酸序列、蛋白质的结构/辅助数据和与蛋白质相关的噪声(图1a所示)。突变生成器与两个鉴别器和亲和力预测器一起,在新的两阶段对抗训练过程中组织,使突变生成器能够提取所需的特征。得益于其架构,突变生成器能够确定突变位点和这些位点上的氨基酸替换(图1b所示),从而朝向指定的亲和力变化方向(图1c所示)。
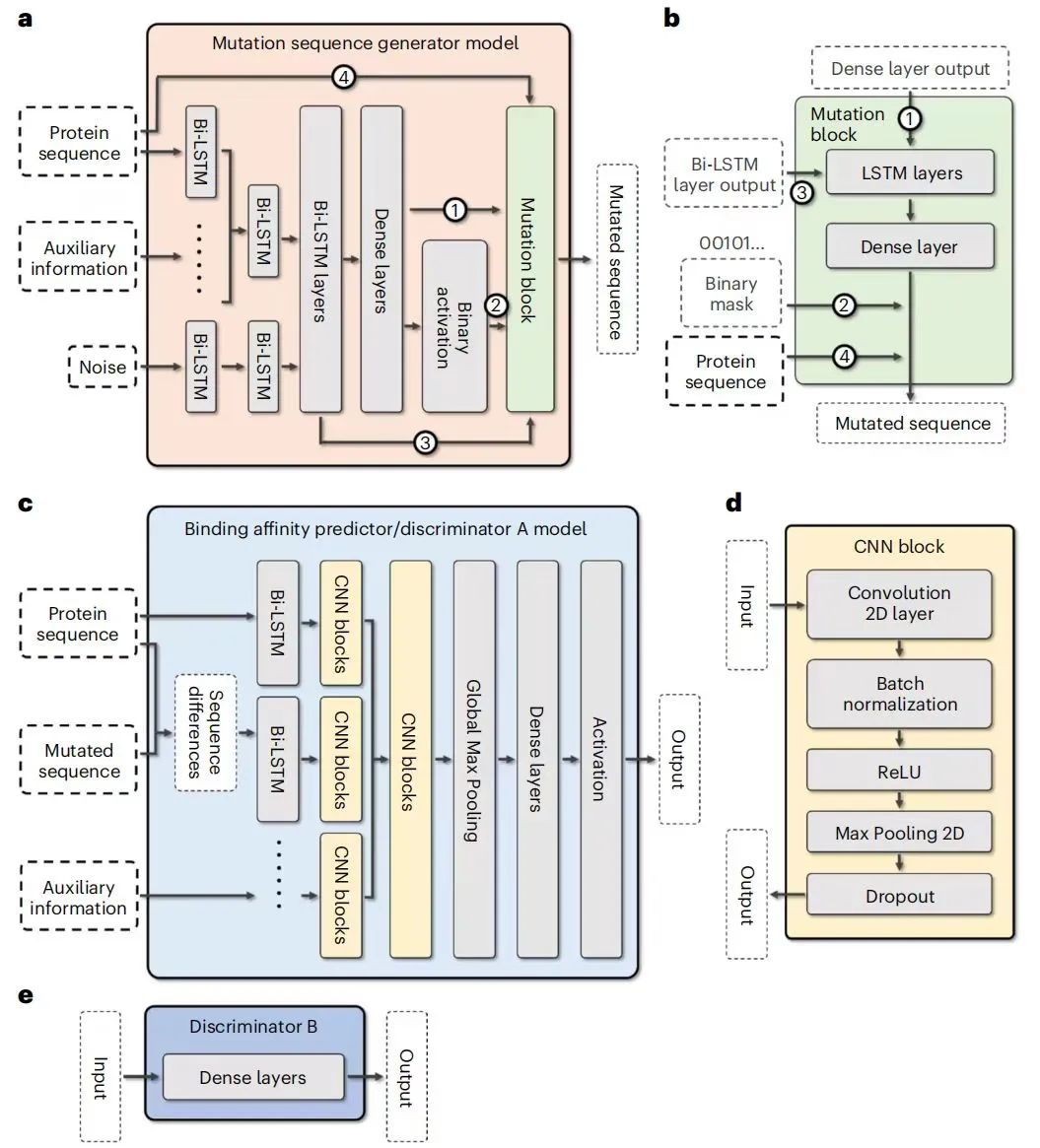
DeepDirect架构(图2)
研究展示了DeepDirect在多个案例中的应用,包括Novavax疫苗构建的优化和SARS-CoV-2 Omicron刺突蛋白的进化路径分析。结果显示,DeepDirect在多个案例中的应用成功展示了其优越性。在Novavax疫苗构建优化和SARS-CoV-2 Omicron刺突蛋白的进化路径分析中,DeepDirect生成的突变序列与随机突变相比,在体外显示出超过一个数量级的结合自由能变化。特别是在Novavax–血管紧张素转换酶相关羧肽酶疫苗构建的案例研究中,展示了其生成更强复合体的能力。
DeepDirect方法在解决蛋白质亲和力方向显著变化的突变生成问题中具有有效性和创新性,通过对抗学习过程动态标记蛋白质数据的“真实”侧并生成伪数据,构建了一个深度学习架构,有效地指导了突变的产生。这一方法不仅在蛋白质生物工程、药物设计、抗体重新配方和治疗性蛋白质药物等领域展示了巨大的应用潜力,也为理解生物分子间的相互作用和蛋白质进化提供了新的视角。
该研究中,李金艳研究员为通讯作者;原悉尼科技大学计算机科学系、现澳大利亚Natexl公司的CEO Tian Lan博士为一作;悉尼科技大学计算机科学系的博士生Shuquan Su、Pengyao Ping,悉尼科技大学生物医学工程系的Grorgy Hutvagner教授,新南威尔斯大学儿童癌症研究院的Tao Liu副教授,深圳理工大学(筹)计算机科学与控制工程学院潘毅教授作为合作作者参与该项研究工作。





